近日,磁共振中心副研究员龚洲和研究员唐淳发展建立了一种基于化学交联质谱分析研究蛋白质结构和动态变化的新方法,相关研究成果发表在期刊《Structure》上。
对蛋白质的动态结构的解析能揭示蛋白质行使功能的内在机制。化学交联质谱分析(Chemical cross-linking coupled with mass spectrometry,CXMS),结合高灵敏质谱分析,是近十年发展起来的结构生物学技术。利用有特定长度的化学交联剂将蛋白质中的特定氨基酸连接起来,就可获得蛋白质上特定氨基酸之间的距离信息。CXMS技术具有灵敏度高,不受分子量大小限制等特点,正受到越来越广泛的青睐和使用。
CXMS测量所获得的距离约束传统上施加于蛋白质的Cα原子或Cβ原子之间,这样的距离约束会超过20![]() ,因此对结构精度的提升有限。针对这一问题,研究团队通过对已有的CXMS实验数据进行比对分析,同时借鉴生物磁共振的思路,发展了将交联剂修饰的侧链Cω原子作为距离约束点的新方法。这种方法将约束范围缩小到了6
,因此对结构精度的提升有限。针对这一问题,研究团队通过对已有的CXMS实验数据进行比对分析,同时借鉴生物磁共振的思路,发展了将交联剂修饰的侧链Cω原子作为距离约束点的新方法。这种方法将约束范围缩小到了6![]() 以内,就大大提高了蛋白质结构计算的精度。这种距离约束不仅提高了蛋白质的静态结构的计算精度,还可以分析表征蛋白质结构的动态变化。
以内,就大大提高了蛋白质结构计算的精度。这种距离约束不仅提高了蛋白质的静态结构的计算精度,还可以分析表征蛋白质结构的动态变化。
龚洲为论文的第一作者,唐淳为通讯作者,联合培养的博士生叶尚祥参与了部分研究工作。该项研究得到了科技部和国家自然科学基金委的资助。
研究团队长期致力于生物大分子动态学的新方法发展与应用研究。在基于化学交联质谱的计算分析领域取得了一系列的研究成果,包括发展了研究蛋白质复合体动态结构的交联计算方法(Biophys Rep, 2015,1:127-138),发展了基于过长交联对信息揭示蛋白质瞬态构象的研究方法(J Biol Chem, 2017,292:1187-1196),整合化学交联质谱分析,小角X射线和单分子荧光表征蛋白质动态结构的新方法(Biochemistry, 2018, 57:305-313)。近期还针对化学交联距离的分布特征进行了系统研究,提出了更加精确的交联距离分布模式(J Phys Chem B, 2020, 123:4446-4453)。
今年发表的两篇论文的主要工作均在今年2月-4月疫情期间完成的。武汉封城,实验室关闭,研究团队采取远程办公的方式,完成了数据分析,结构建模,计算模拟等工作。同时还基于网络会议软件开展交流讨论,保持了“停班不停工”的良好状态”。
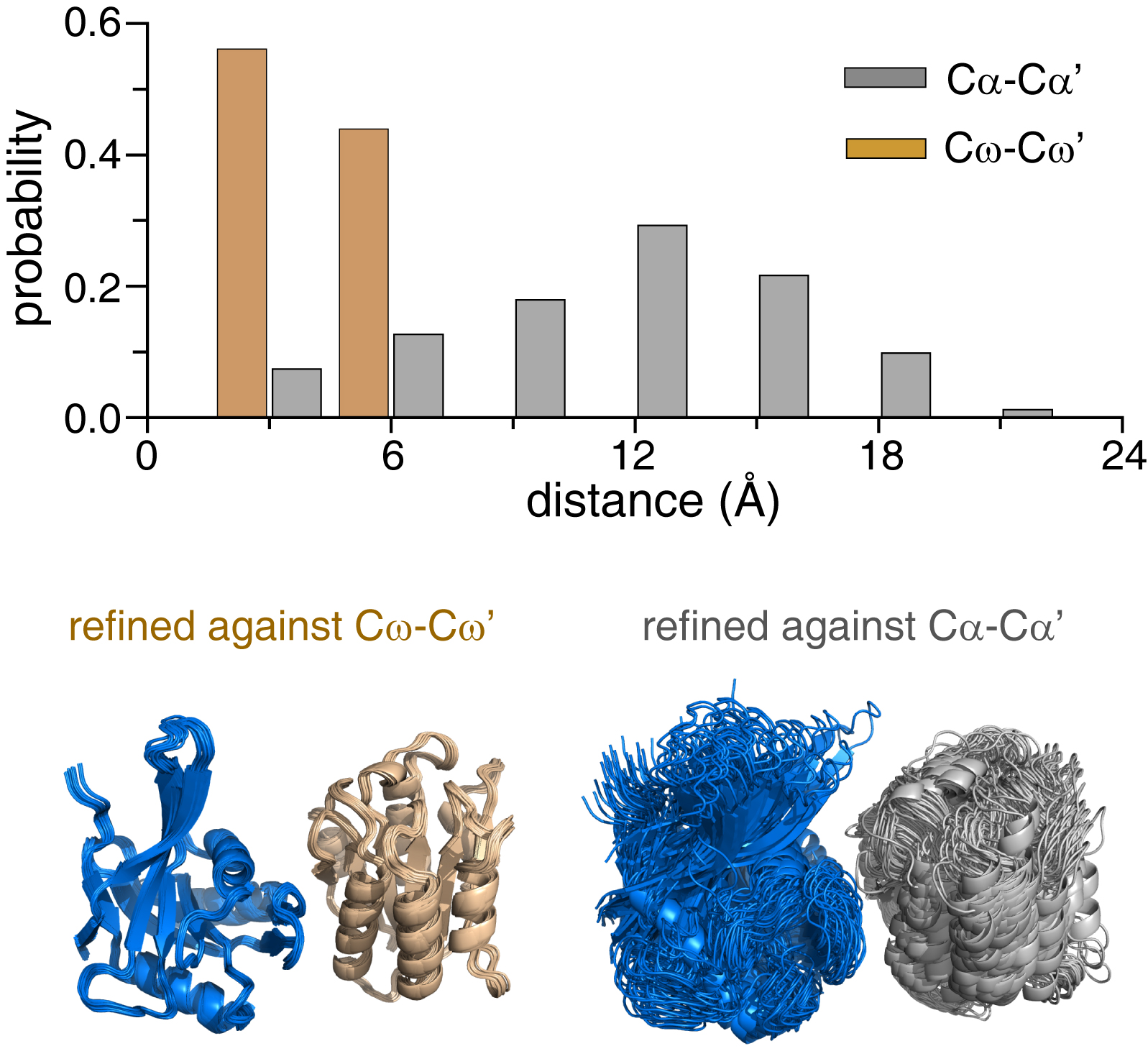
新的交联约束方式大幅度提高蛋白质结构计算的精度